
Combiner simulations moléculaires et RMN pour étudier les électrolytes
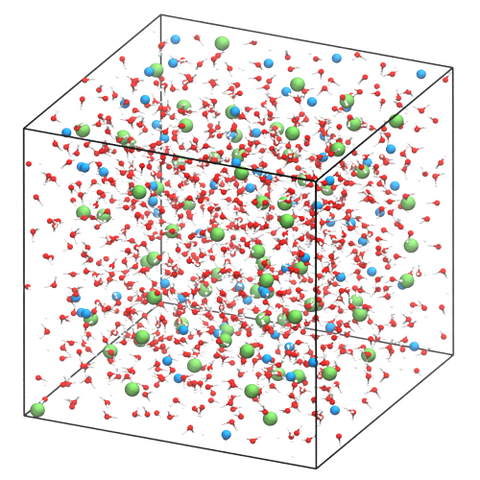
Dans le cadre d’une collaboration franco-américaine, des chercheurs du laboratoire PHENIX ont récemment proposé de combiner des calculs ab initio à des simulations de dynamique moléculaire classique pour interpréter des données de relaxation RMN obtenues par le laboratoire américain pour l’ion Na+ en solution dans l’eau. La compréhension de la solvatation des ions Na+ présente en effet un intérêt considérable pour étudier la dynamique de systèmes physiologiques comme les cellules et les tissus biologiques, ou les électrolytes utilisés dans les dispositifs de stockage électrochimique de l’énergie.
Si la spectroscopie de résonance magnétique nucléaire (RMN) est utilisée en routine pour déterminer la structure des molécules, son potentiel dépasse largement cette caractérisation. Des dispositifs expérimentaux de plus en plus performants et une maîtrise approfondie des principes de cette technique permettent en effet d’étudier la dynamique microscopique autour des noyaux des atomes, de suivre « en direct » une réaction chimique ou encore de mesurer des coefficients de diffusion dans différents milieux.
L’interprétation des données dynamiques recueillies, comme la relaxation de l’aimantation macroscopique d’un échantillon suite à son excitation par un champ magnétique extérieur, nécessite cependant des modèles microscopiques réalistes pour remonter au processus dynamique que l’on cherche à élucider. Dans le cadre d’une collaboration franco-américaine, des chercheurs du laboratoire PHENIX ont récemment proposé de combiner des calculs ab initio à des simulations de dynamique moléculaire classique pour interpréter des données de relaxation RMN obtenues par le laboratoire américain pour l’ion Na+ en solution dans l’eau.
La compréhension de la solvatation des ions Na+ présente en effet un intérêt considérable pour étudier la dynamique de systèmes physiologiques comme les cellules et les tissus biologiques, ou les électrolytes utilisés dans les dispositifs de stockage électrochimique de l’énergie. Ces simulations prédisent correctement les temps de relaxation RMN observés expérimentalement sur une large gamme de températures et de concentrations en sel et montrent que les modèles simples utilisés jusqu’ici dans la littérature ne sont pas adaptés. La relaxation RMN de l’ion Na+ est due essentiellement à la dynamique collective des molécules d’eau dans les deux premières couches de solvatation à une échelle de temps très courte.
Les effets de la température et de la concentration en sel s’expliquent quant à eux par l’évolution globale de la dynamique de la solution à des échelles de temps plus longues. Ces résultats*, présentés dans la revue Nature Communications, démontrent que combiner théorie et expériences de relaxation RMN s’avère être un outil puissant pour développer une meilleure compréhension de la dynamique des systèmes (bio-)électrolytiques.
*Ces recherches s’inscrivent dans le projet ERC SENSES (making Sense of Electrical Noise by Simulating Electrolyte Solutions)
Chubak, I., Alon, L., Silletta, E.V. et al. Quadrupolar 23Na+ NMR relaxation as a probe of subpicosecond collective dynamics in aqueous electrolyte solutions. Nat Commun 14, 84 (2023). https://doi.org/10.1038/s41467-022-35695-3
Contact
Benjamin Rotenberg
Chercheur au laboratoire Physicochimie des électrolytes et nanosystèmes interfaciaux (CNRS/Sorbonne Université)
