
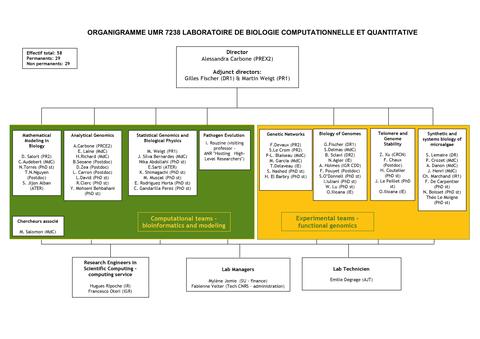
Laboratoire de biologie computationnelle et quantitative | LCQB
-
Unité mixte de recherche
Le LCQB est un laboratoire interdisciplinaire travaillant à l'interface entre la biologie et les sciences quantitatives. Il est construit pour promouvoir une interaction équilibrée des approches théoriques et expérimentales en biologie et pour favoriser la définition de nouvelles questions expérimentales, l'analyse des données et la modélisation des phénomènes biologiques. Nos projets abordent des questions sur les structures et processus biologiques à travers la réalisation d’expériences de génomique fonctionnelle, d’ingénierie des génome et de biologie synthétique sur des organismes modèle (levure, micro-algue et bactérie), le séquençage des génomes, la génération in silico de nouvelles données biologiques qui restent aujourd'hui inaccessibles aux expériences, la modélisation mathématique et physique des systèmes biologiques, le développement de méthodes statistiques pour les données l'analyse, et la conception d'algorithmes originaux visant à des prédictions.
Équipes
- Analytical Genomics (Group Leader: Alessandra Carbone)
-
Biology of Genomes (Group Leader: Gilles Fischer
-
Evolution and immunology of pathogens (Group Leader: Igor Rouzine)
-
Genetic Networks (Group Leader : Frederic Devaux)
-
Mathematical Modeling in Biology (Group Leader: Delphine Salort)
-
Statistical Genomics and Biological Physics (Group Leader: Martin Weigt)
-
Synthetic and Systems Biology of Microalgae (Group Leader: Stéphane Lemair)
-
Telomere & Genome Stability (Group Leader: Zhou Xu)
Direction
-
Alessandra Carbone
Direction adjointe
-
Gilles Fischer
-
Martin Weigt
Administration
-
Mylène Jomie
-
+33 1 44 27 73 38
-
Fabienne Velter
-
+33 1 44 27 20 67
Communication
-
Fabienne Velter
-
+33 1 44 27 20 67
Tutelle hébergeante
- Sorbonne Université
Autres tutelles
- Centre National de la Recherche Scientifique | CNRS
58 membres
-
Enseignants-chercheurs : 18
-
Chercheurs : 4
-
Personnels d'appui à la recherche : 1
-
Doctorants : 19
-
Post-doctorants : 7
- Microfluidic single cell microscopy
- OXford Nanopore Sequencing
- Pulse-field gel electrophoresis
- Microdissecator
- Microalga culture room
- Computer cluster
- Logiciels
Publications
Publications sur HAL - Archive ouverte
Toutes les publications sur HALAdipocyte size distribution: Mathematical model of a tissue property
Aloïs Dauger, Hedi Soula, Chloe Audebert
Mathematical Biosciences, 2025, 384, pp.109433. ⟨10.1016/j.mbs.2025.109433⟩. ⟨hal-05042738⟩
Integrating Machine Consciousness Simulation and LLMs Toward Verbal and Non-Verbal General Intelligence in Artificial Agents
Tonglin Yan, Grégoire Sergeant-Perthuis, Kenneth Williford, David Rudrauf
2025. ⟨hal-05064693⟩
PCM-LLMS: A HYBRID ARCHITECTURE TO ENHANCE HUMAN-LIKE SOCIAL INTELLIGENCE IN VIRTUAL AGENTS
Tonglin Yan, Grégoire Sergeant-Perthuis, David Rudrauf
2025. ⟨hal-05054281⟩
Structure-Function analysis of the yeast mitofusin Fzo1
Laetitia Cavellini, Mohammad Ozeir, Maria de los Ángeles Morcillo Parra, Yan Tang, Éric Vandjour, Thomas Chenuel, Julien Henri, Philippe Meyer, Mickael M. Cohen
Mitochondria Metabolism And Signaling, Mar 2025, Ventura (California), United States. ⟨hal-05019697⟩
Contact details
Adresse physique
75005 Paris
Adresse postale
75005 PARIS
-
+33 1 44 27 42 52
Institut de Biologie Paris Seine
Sorbonne Université
Bâtiment C, 3ème et 4ème étages
7-9 Quai Saint Bernard 75005 Paris
